配文摘要
AggreProt是一个用于预测和设计蛋白质聚集倾向区域的在线工具,自动利用深度神经网络的集合来预测蛋白质序列中的聚集易感区域(APRs)。该服务器通过交互式序列和结构查看器提供了一个直观的界面,提供了每个残基的聚合概况,以及关于溶剂可及性和跨膜倾向的信息,用于综合分析。它在指导蛋白质工程策略以降低聚集倾向和提高溶解度方面的具有很大潜力。
背景
重组蛋白在包括工业生物催化剂或疗法在内的许多应用中发挥着关键作用。尽管最近计算蛋白质结构预测取得了进展,但蛋白质的溶解度和聚集性降低仍然是设计的挑战。识别易发生聚集的区域对于理解错误折叠疾病或设计高效的蛋白质技术至关重要,因此具有巨大的社会经济影响。

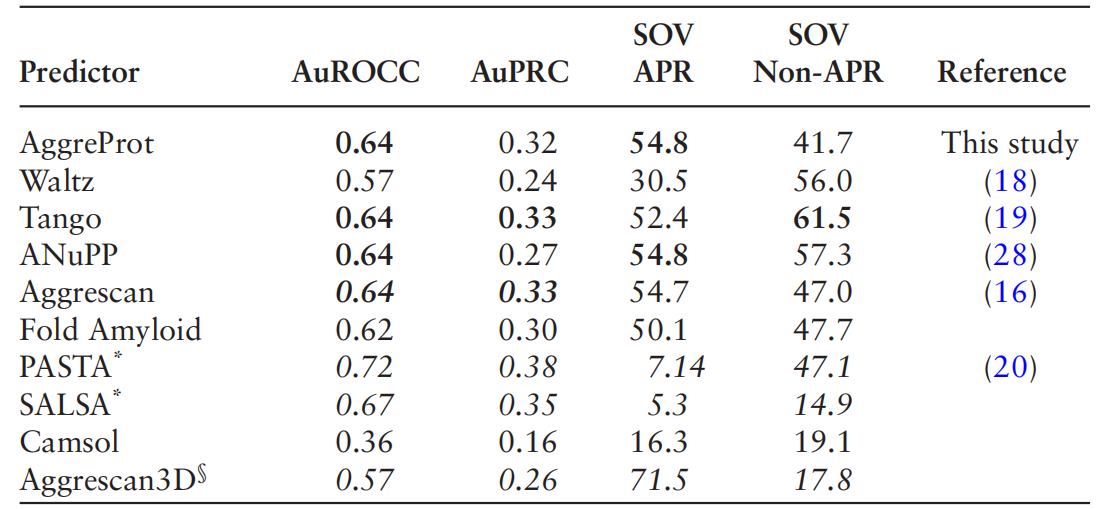
2024年5月27日丹麦研究人员在Nucleic Acids Research上发表了一篇名为“AggreProt: a web server for predicting and engineering aggregation pron eregions in proteins”的文章。文章介绍了AggreProt:一个用于预测和设计蛋白质聚集倾向区域的web服务器。自动利用深度神经网络的集合来预测蛋白质序列中的聚集易感区域(APRs)。经过实验评估的六肽训练,AggreProt在两个独立的基准数据集上比较或优于最先进的算法。该服务器通过交互式序列和结构查看器提供了一个直观的界面,提供了每个残基的聚合概况,以及关于溶剂可及性和跨膜倾向的信息,用于综合分析。研究人员在几个用例中证明了AggreProt在预测蛋白质的不同聚集行为方面的有效性,这强调了它在指导蛋白质工程策略以降低聚集倾向和提高溶解度方面的潜力。该web服务器可在https://loschmidt.chemi.muni.cz/aggreprot/免费获取。
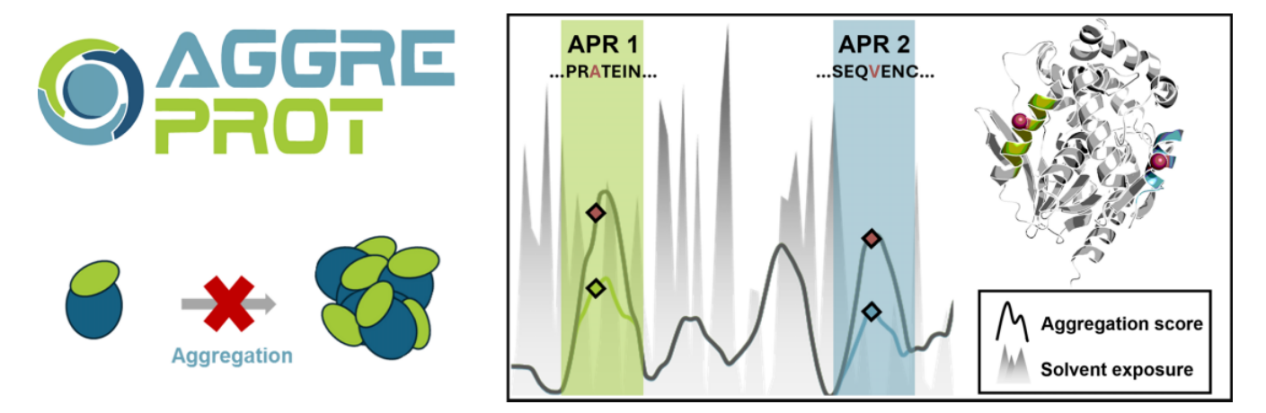
蛋白表达现状问题
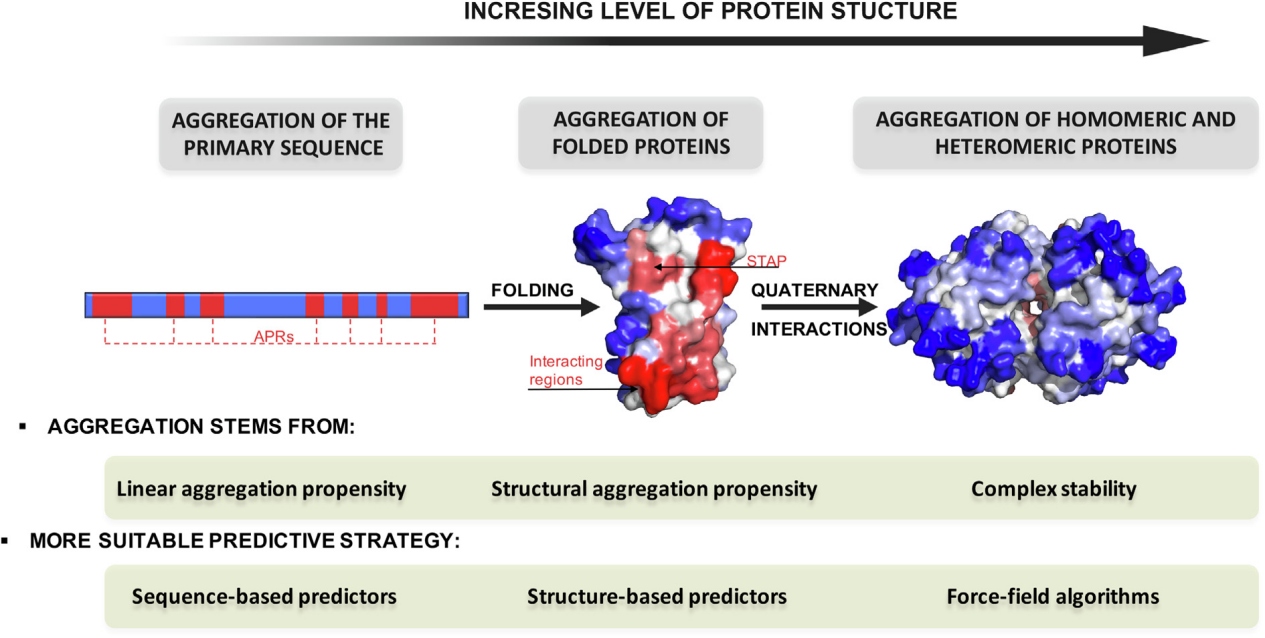
重组蛋白在包括工业生物催化剂或疗法在内的许多应用中发挥着关键作用,但是包涵体形成,低纯化蛋白产量,聚集/沉淀是蛋白表达常见的问题。蛋白质折叠主要由疏水残基的埋藏驱动,疏水残基的暴露可导致非天然的自结合、错误折叠和最终的聚集。这种错误折叠聚集体的形成可由多种因素触发,并与阿尔茨海默氏病或帕金森病等严重疾病相关。在聚集物中,淀粉样蛋白代表了一种以高度组织化的二维结构为特征的特殊类别。淀粉样蛋白是由堆叠的蛋白质分子重复单元形成的,通过其交叉-β-片层结构内的氢键分子间网络稳定,然而,这些分子通常采用不同的形态。它们有一个共同的结构内核,该结构内核被认为是淀粉样蛋白形成的重要驱动因素,并对其稳定性至关重要。因此,这些区域(APRs)是设计突变的完美靶标,这些突变可以减少聚集倾向,从而提高蛋白质的溶解度。
已经设计了几种算法来解决聚集问题。根据它们接受的输入数据类型,这些算法被分类为顺序或结构预测。这两个类别的算法极大地促进了我们在分子水平上对蛋白质聚集和溶解度的理解,并且经常被用于识别蛋白质中的APRs,并取得了不同程度的成功。
(数据来源SantosJ, et al. Comput Struct BiotechnolJ. 2020)
在过去几年中,基于机器学习的第三代预测期也已经出现,例如淀粉蛋白预测器中的支持向量机,RF淀粉样蛋白和Amylogram中的随机森林分类器,以及许多其他包括ANuPP,FishAmy-loid或CORDAX。

(数据来源Prabakaran R,et al. J Mol Biol. 2021)
AggreProt的特点优势
AggreProt可以预测整个蛋白质序列的聚集趋势,而不仅仅是单独的六肽片段。与其他基于序列或机器学习的最先进方法相比,AggreProt在残基级别或SOV验证方面达到了相似或更好的评估性能。
AggreProtweb服务器的使用流程
该服务器将其专用的淀粉样蛋白聚集倾向预测器与跨膜(TM)倾向和溶剂可及表面积(SASA)计算相结合,为分析的蛋白质序列提供结构背景。
数据输入:用户以FASTA格式输入蛋白质序列,服务器会快速检查序列的完整性,包括头部和序列是否存在。该服务器允许同时输入多达三种不同的蛋白序列。用户可以上传与输入序列相关的结构文件(接受PDB和mmCIF格式)。如果用户不能提供结构文件,AggreProt提供了从AlphaFoldDB获取结构的选项。
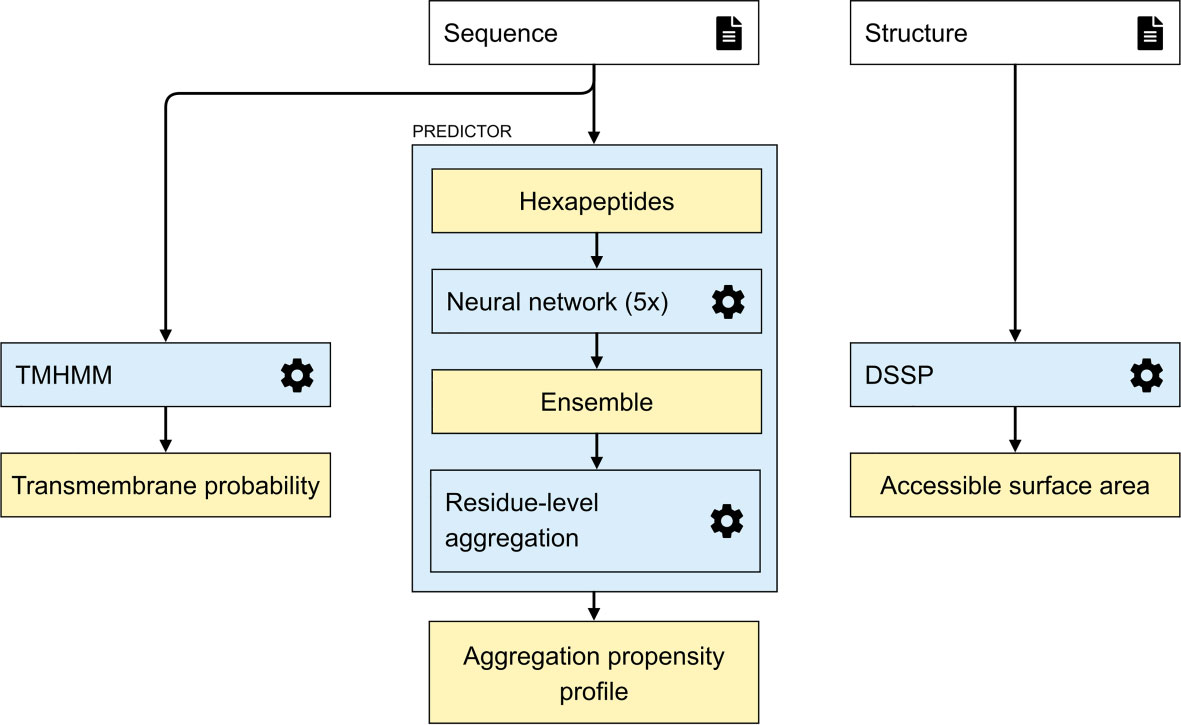
结果输出:完成计算后,作业状态会变为“完成”,结果以图形形式展示,包括对齐的轮廓图和序列显示图。用户可以通过调整阈值来改变聚集倾向的敏感性和特异性。聚集倾向曲线用半透明的实色相表示,TM倾向和SASA分别用点和虚线表示。在可视化工具(倾向图)的这一部分中,将鼠标悬停在任何序列位置上,就会显示有关蛋白质残基的附加信息以及计算出的每种倾向的单个预测值。提供交互式的序列和结构查看器,允许用户比较多个蛋白质的轮廓图。如果用户提供了蛋白质的三维结构,或者从数据库中获取了结构,AggreProt会提供一个交互式的三维视图。

案例验证
AggreProt被用来识别HLDLinB中的聚集倾向区域(APRs),基于AggreProt的预测结果,研究人员设计了一系列突变,目的是降低HLDLinB的聚集倾向并提高其溶解度。对HLDLinB设计的突变进行了实验评估,结果显示AggreProt能够正确识别出可以减少聚集倾向并增加可溶蛋白产量的突变。并且发现AggreProt和其他预测器相比检测LinB中APRs的能力要强。
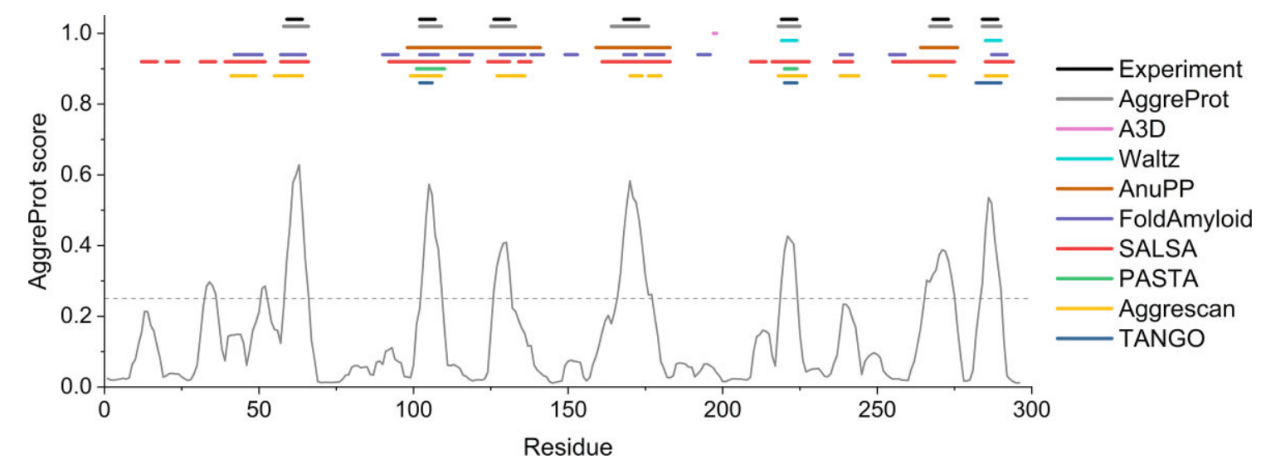
使用SoluProtMutDB数据库中的深突变扫描数据,分析了III型聚酮合酶和TEMβ-内酰胺酶。发现许多增加溶解度的突变与AggreProt预测的APRs相对应。实验观察到的突变效果与AggreProt预测的效果在多个案例中一致,证实了AggreProt的有效性。对于暴露于蛋白质表面的APRs,AggreProt能够较好地预测突变对聚集倾向的影响,对于埋藏在蛋白质内部的APRs,预测突变的影响则更为复杂。如果突变增加了局部疏水性,AggreProt可能会预测增加聚集倾向,这与暴露APRs的预测相反。AggreProt的预测准确性与突变的性质紧密相关,包括突变的类型、位置(表面或埋藏)、以及对蛋白质结构和溶剂可及性的影响。

总结与展望
AggreProt是一个基于深度神经网络的web服务器,可以预测蛋白质序列中的聚集易发区域(APRs),AggreProt在两个独立的基准数据集上的性能与或优于现有的最先进算法。AggreProt提供了直观的界面,包括序列和结构可视化,以及聚集倾向、跨膜倾向和溶剂可及性的分析。
AggreProt在预测蛋白质突变对聚集行为的影响方面表现良好,可以为蛋白质工程提供指导。由于降低蛋白质聚集倾向的最终目标只能通过对其序列进行改造来实现。未来研究人员会增强用户界面:提供一个“设计”面板,使用户能够规划和预测蛋白质工程策略的结果。允许用户微调APRs的边界,定义自定义区域,以更精确地实施工程策略。实现针对APRs的特定突变策略,如守门残基的替代和暴露残基的饱和突变。提供更多自定义选项,让用户能够选择和组合不同的突变策略。展示经过AggreProt评估后的理想多突变序列,供用户检查和使用。通过继续提升AggreProt的性能,使其成为一个更加强大的蛋白质工程工具。
