TurboID是一种工程化的生物素连接酶,它利用ATP将生物素转化为生物素-AMP,这是一种活性中间产物,可共价标记邻近的蛋白质。通过定向进化进行优化,TurboID的活性比之前描述的与生物素连接酶相关的邻近标记方法(如BioID)高得多,从而能够实现更高的时间分辨率和更广泛的应用。split-TurboID由TurboID的两个非活性片段组成,它们可以通过蛋白质-蛋白质相互作用或细胞器-细胞器相互作用重新组装,这有助于提高比单独的全长酶更高的靶向特异性。通过TurboID或split-TurboID进行生物素化的蛋白质随后用链霉亲和素珠进行富集,并通过质谱法进行鉴定。

2020年11月研究人员在Nat Protoc上发表了一篇名为“Proximity labeling in mammalian cells with TurboID and split-TurboID”的文章,这是一篇关于TurboID和split-TurboID在哺乳细胞中邻近标记的protocal。作者概述如何使用实验室最新的PL酶TurboID6和split-TurboID进行亚细胞蛋白质组学图谱的绘制。作者详细描述了如何进行融合结构设计和表征(可变时间),蛋白质组样品制备(5-7d),质谱数据获取(2d)和蛋白质组数据分析(1周)。
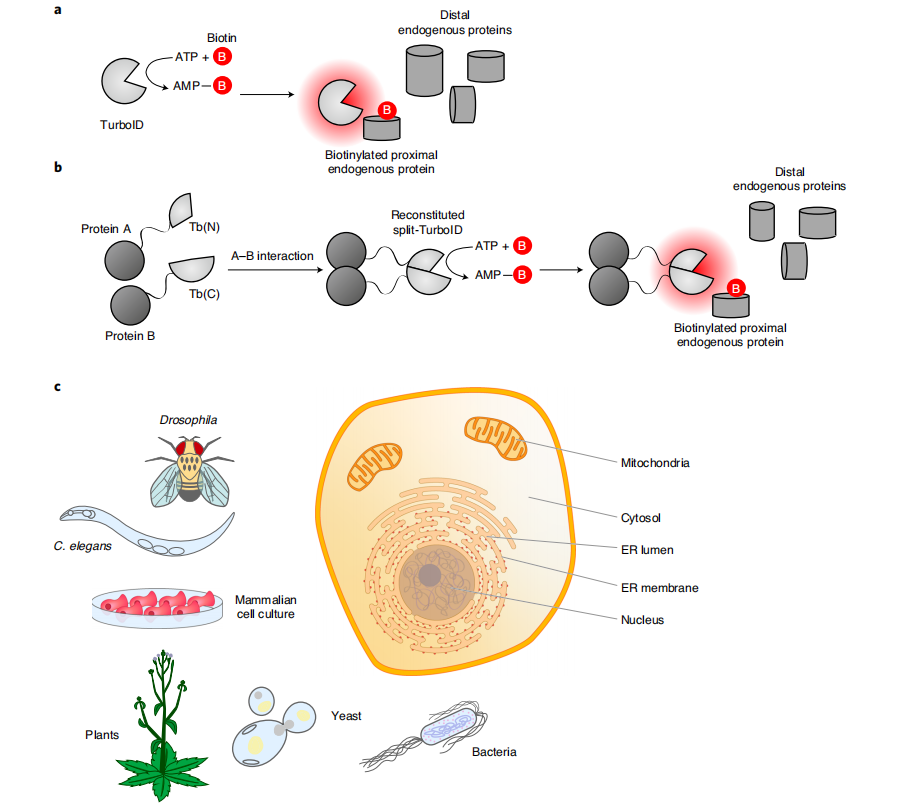
TurboID邻近标记技术的局限性
不同的亚细胞区域具有不同的PH,氧化还原环境,内源性核苷酸浓度,导致TurboID的活性存在差异。TurboID的活性差异也因细胞器和生物体类型不同而存在差异;并且它与蛋白融合可能会影响蛋白质的稳定性,定位,功能;因此评估拟用于蛋白质组学的TurboID融合构建体的活性至关重要。这种分析法可能无法考虑双重定位的蛋白质,导致覆盖率降低(例如,一种同时存在于ERM和细胞质中的蛋白质会在使用细胞质TurboID作为参考的第二轮筛选步骤中被去除。)
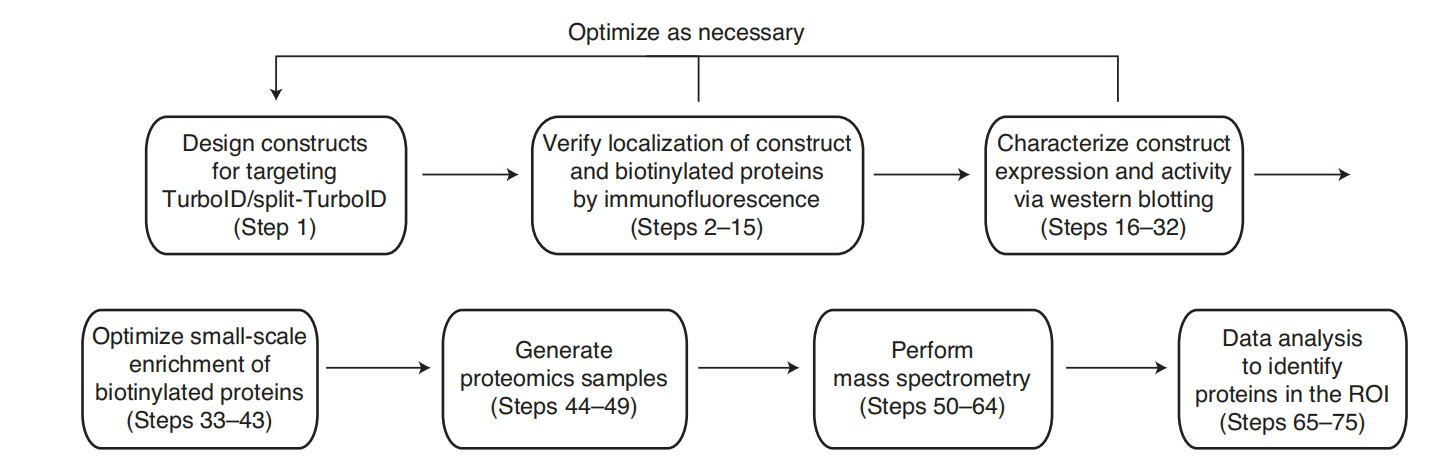
TurboID蛋白质组学实验的流程概述
进行TurboID蛋白质组学实验的工作流程:设计的融合构建体应使用免疫荧光和蛋白质印迹法进行表征,并在必要时进行优化。在构建体验证之后,在生成用于质谱分析的蛋白质组样本之前,需要对富集条件进行优化。最后在质谱分析后进行数据分析已识别富集的蛋白。数据分析主要使用比率法结合基于ROC的阈值分析。
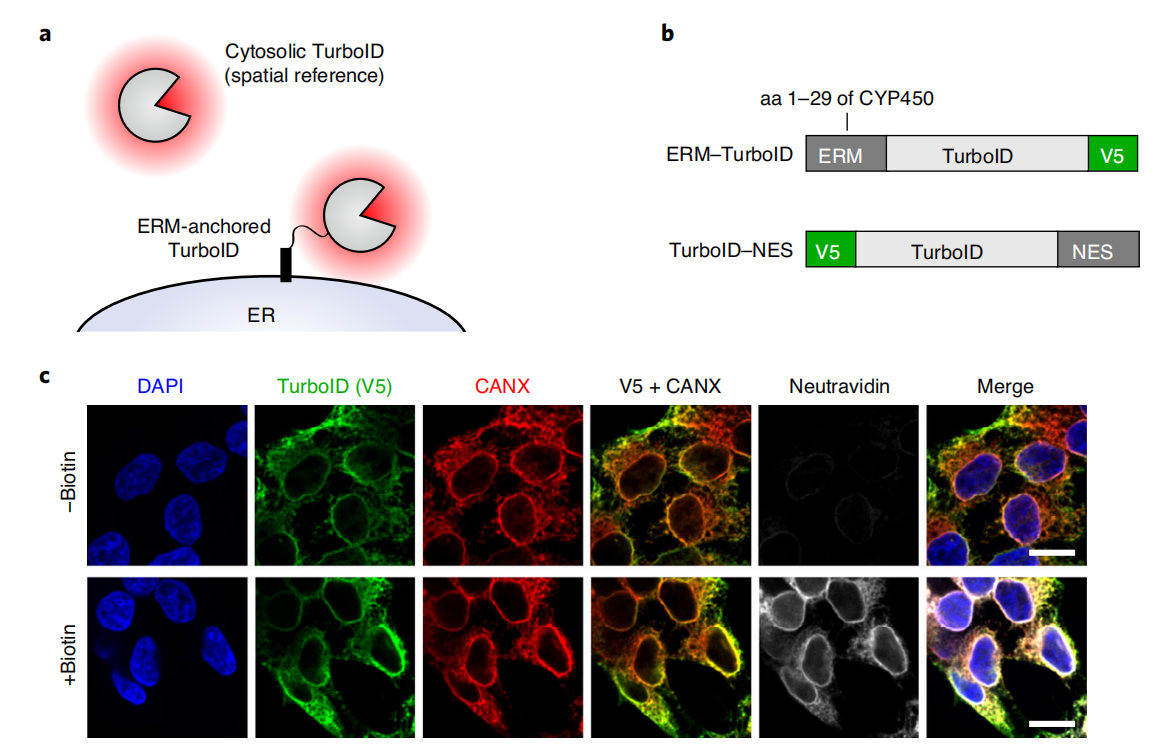
将TurboID定位到目标区域
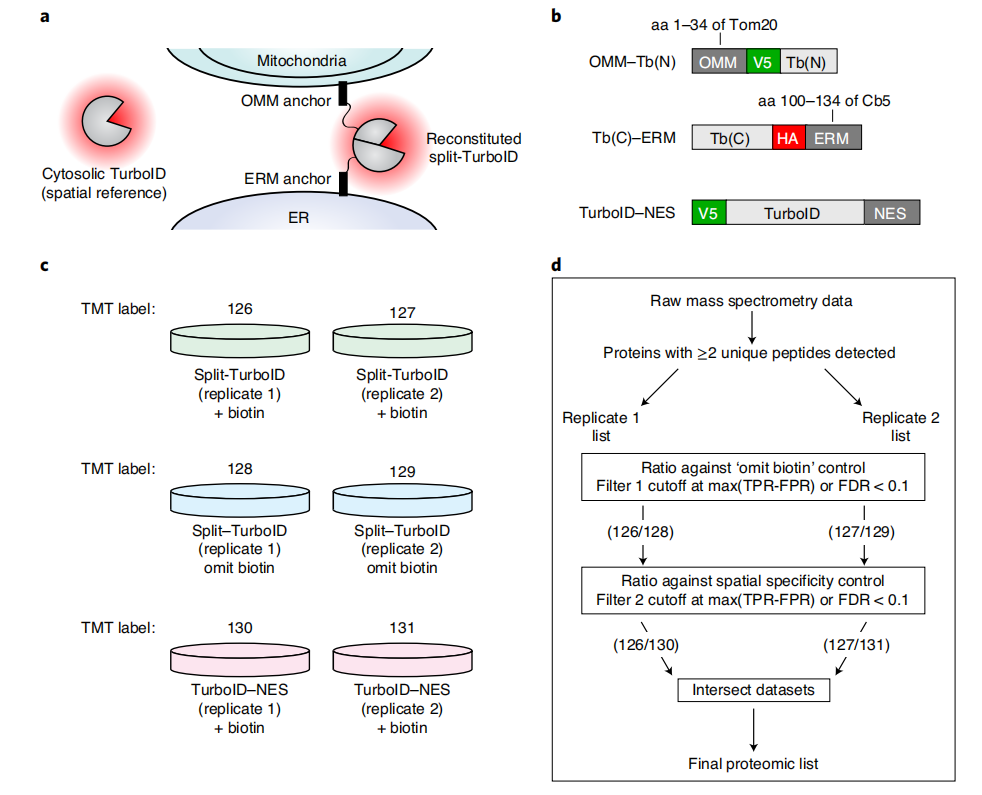
首先需要将构建体靶向到感兴趣的细胞区域或蛋白质复合物。对于细胞器或亚细胞结构的蛋白质组图谱绘制,可以通过使用定位序列来实现靶向。使用从细胞色素P450和细胞色素b5衍生的靶向肽将TurboID和split-TurboID靶向到细胞外基质ERM并使用从线粒体抗病毒信号蛋白(MAVS)和外膜20千道尔顿亚基转位酶(Tom20)衍生的靶向肽将其靶向到外膜(OMM)。通过免疫染色并与针对相同细胞器的共转染/感染的荧光蛋白标记物进行比较,或者通过与靶向区域的端源性标记物进行共免疫染色,可以验证构建体的定位。
注意事项:
1)避免通过与全长功能性蛋白融合靶向目标区域,生物活性蛋白的过度表达可能会干扰信号转导和生物学功能。
2)融合后,可能存在独特的亚细胞定位,需要在不同水平下测试多个融合构建载体,以确保正确的定位,将对目标的干扰降低。
3)TurboID或split-TurboID片段被直接融合到正在研究的诱饵蛋白质上。关键是要确保TurboID/split-TurboID片段的融合不会扰乱诱饵蛋白质的定位模式、生物功能或相互作用。
4)确保TurboID和split-TurboID不影响连接酶的活性至关重要。
5)如果构建体未与靶向区域共定位或观察到大量的细胞器扰动,需要对构建体进行优化:改变连接子的长度或刚性,构建体几何形状和靶向序列。通常使用富含甘氨酸和丝氨酸的连接子,起始长度为10个氨基酸。避免使用可能被TurboID标记化学法破坏的FLAG和其他富含赖氨酸的表位标签。TurboID/split-TurboID融合构建体可以通过转染或病毒转导引入哺乳动物细胞。
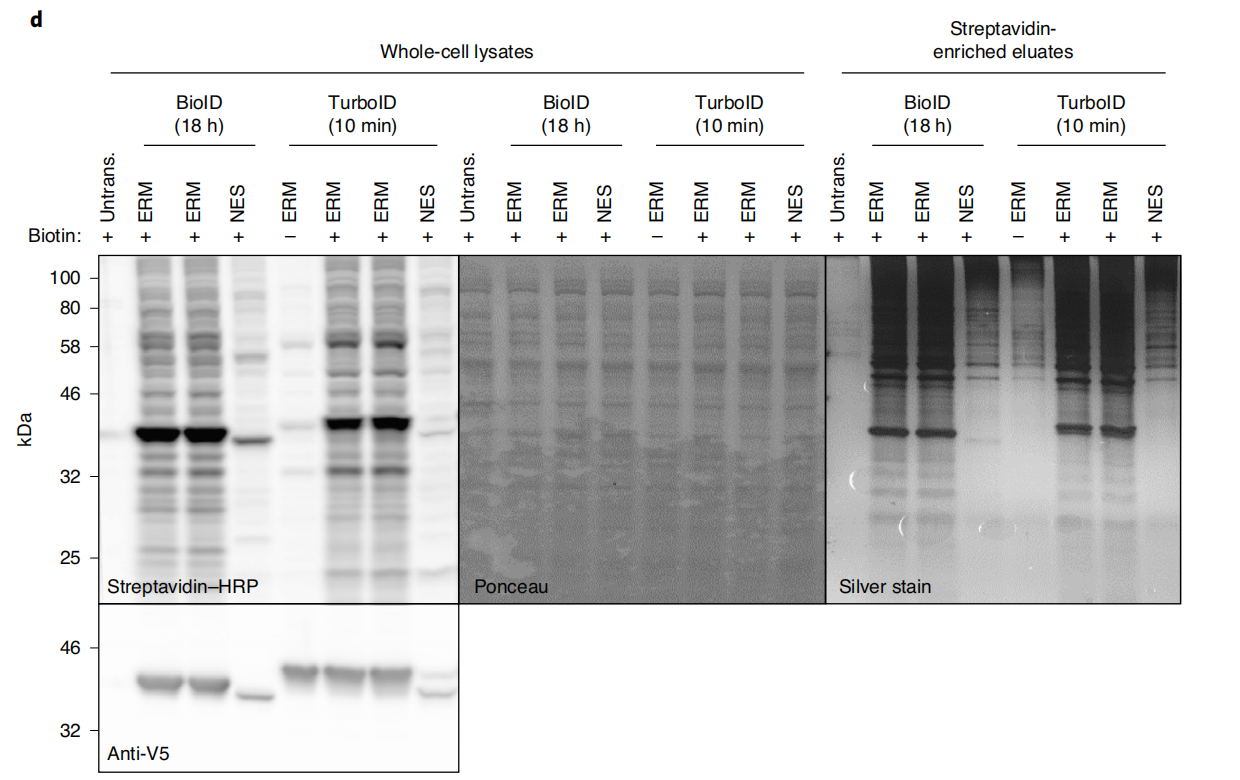
对TurboID构建体的活性进行表征
在验证融合构建体的正确定位和/或功能后,需要评估TurboID/split-TurboID的活性。这可以通过对生物素化蛋白进行免疫荧光染色(例如使用荧光素-亲和素偶联物)或通过全细胞裂解物中生物素化蛋白的蛋白质印迹分析(例如使用链霉亲和素-荧光素或链霉亲和素-HRP偶联物)来完成。还应使用针对相应表位标签的荧光素/HRP抗体偶联物对相同的裂解物进行运行和印迹,以验证TurboID融合构建体的表达和完整性(缺乏蛋白水解酶)。大规模蛋白质组实验前进行小规模的TurboID/split-TurboID生物素化蛋白的富集,通过银染法对洗脱物样本进行分析,以确认实验样本中总蛋白的成功富集,并与阴性对照进行比较。
注意事项:
1)包括不含连接酶或生物素的阴性对照,以评估内源性生物素化蛋白的信号和背景生物素化水平;
2)使用最低的生物素浓度和最短的标记时间,避免非特异性标记;
3)对各种实验条件进行优化,例如,改变磁珠体积,洗涤次数,最大化捕获生物素化物质,并同时最小化非特异性结合到磁珠上;
4)在富集前,使用凝胶过滤柱除去游离的生物素;富集前对内源性生物素化蛋白耗竭,可以增加TurboID/split-TurboID标记蛋白的信号。
蛋白质组学样本制备
从小规模富集中优化的条件可以相应地扩大规模,用于大规模蛋白质组学实验。对于HEK293T细胞中的蛋白质组实验,使用约2000万个细胞的小瓶来生成用于质谱的样本。使用胰蛋白酶或其他水解酶对在链霉亲和素珠子进行酶解消化释放与亲和基质偶练的蛋白质。在去除链霉亲和素珠子后,释放的蛋白质被完全消化成肽段,通过液相色谱-串联质谱法(LC-MS/MS)进行分析。可以使用多种定量和半定量方法来区分实验条件下的蛋白质富集和阴性对照。本实验的的定量法用的是同位素质谱标签法(TMT)。

注意事项:每种实验条件需包含重复样本,包括没有连接酶或生物素的阴性对照,以及空间特异性对照。例如,在使用TurboID对ERM蛋白的分析中或者使用split-TurboID对ER线粒体接触蛋白的分析中,同样使用了包含了TurboID-NES定位于细胞质溶胶的样本以进行比较。
数据分析
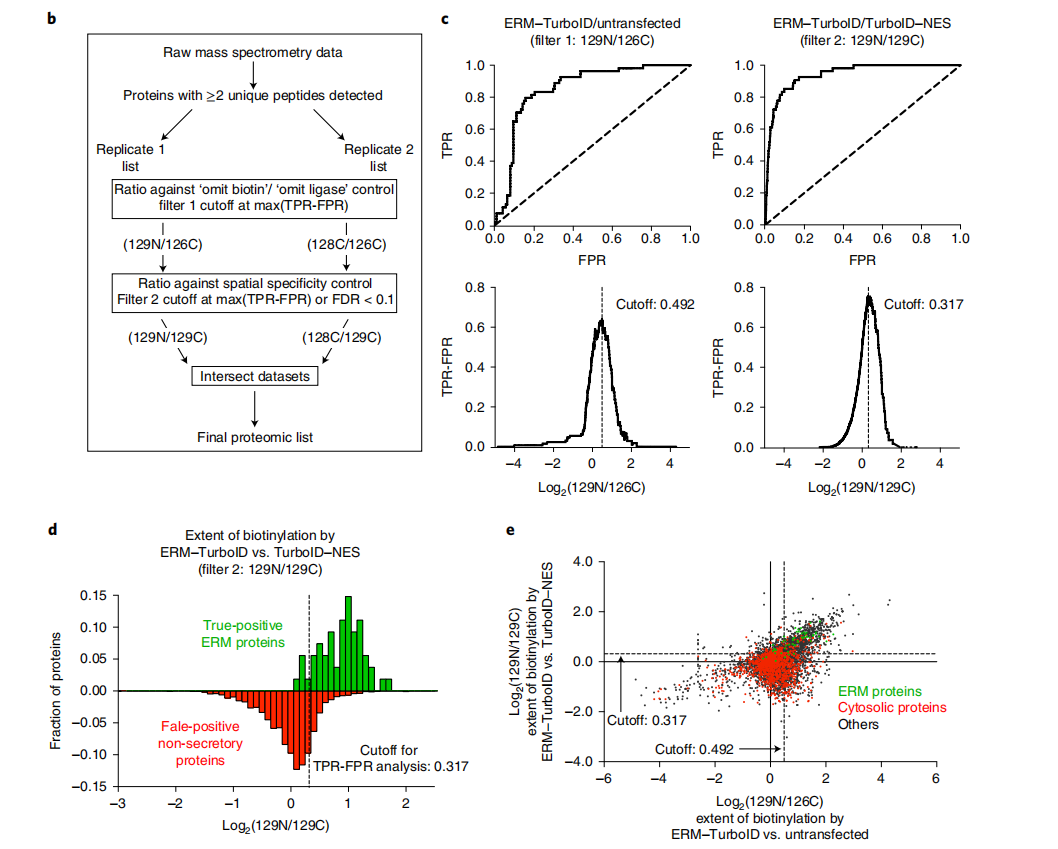
对于定量方法,需要两个基于ROC的筛选步骤:首先确定生物素化蛋白质;然后确定哪些蛋白质优先被针对感兴趣区域的TurboID标记。
通过TMT比率绘制的显示的数据集中真阳性蛋白和假阳性蛋白分布的直方图表明通过ROC确定的截断值成功地富集了真阳性蛋白。高于确定TMT比率截断值的蛋白质被视为生物素化蛋白,而低于截断值的蛋白质则被视为可能的非特异性结合物。第二次筛选的目的是比较实验样本与针对TurboID的参考对照,该对照针对重叠但不同的亚细胞区域(它们位于相邻或连续的亚细胞区域)。通过比较实验样品与参考样品(如细胞质TurboID-NES)的TMT信号,这一步同样需要计算TMT比例,分析确定ROC截断值。进一步过滤蛋白质,以确定哪些蛋白质在目标区域中优先被生物素化。
注意事项:这2个筛选步骤需要重复进行,并独立于每个样本,最终的蛋白质组列表是从每个重复样本的筛选列表的交集生成的。
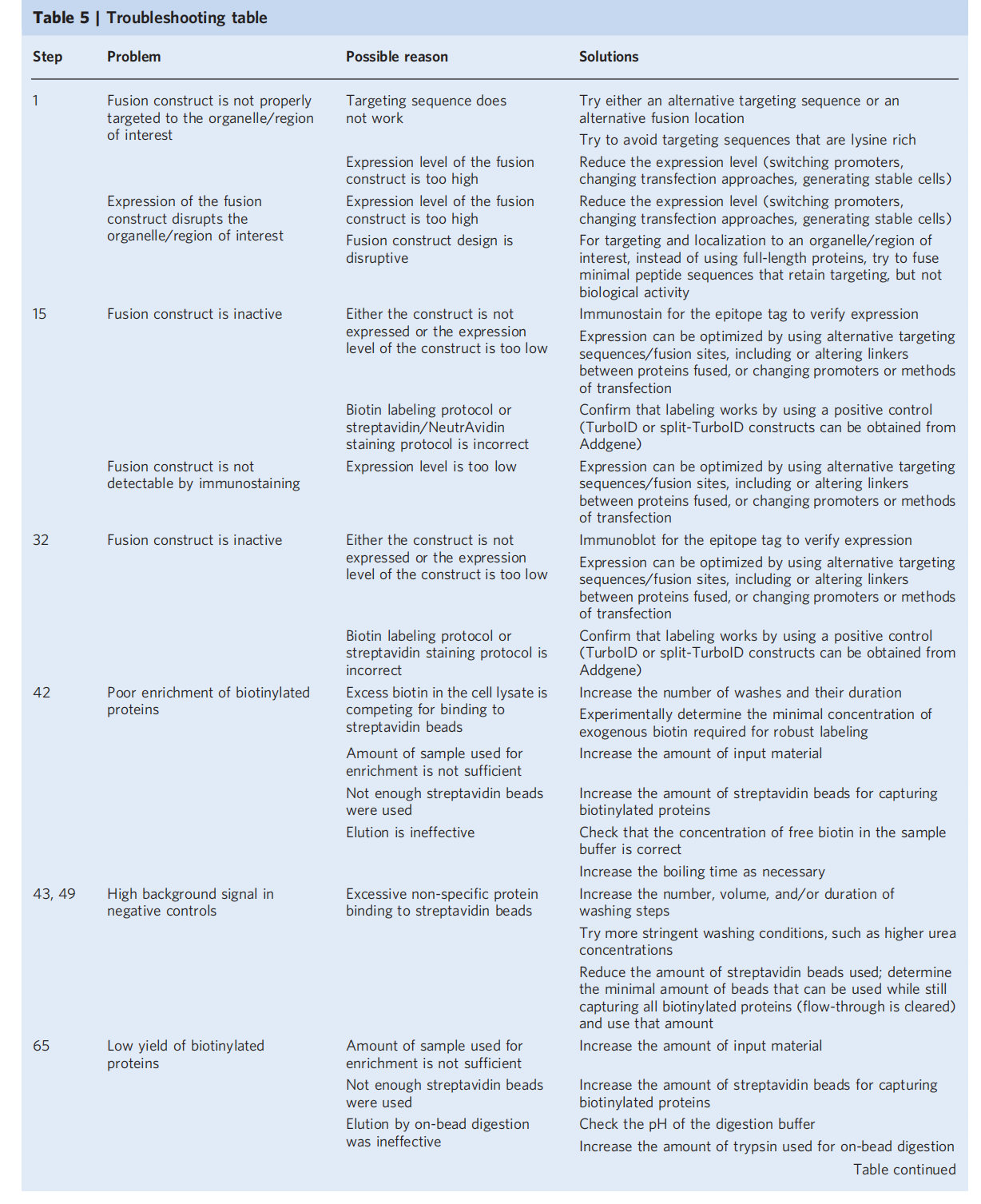
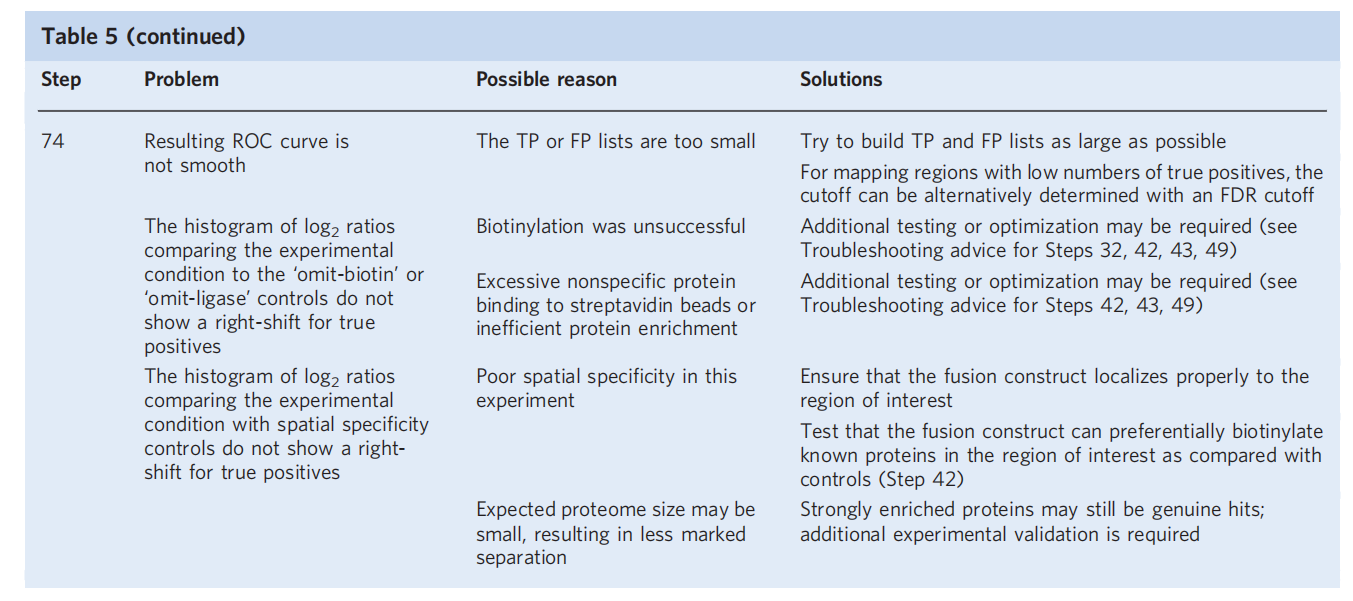
常见问题与解决方案
作者还总结了在使用邻近标记技术时可能会出现的问题以及相应的解决方案。
预期实验结果
针对目标的TurboID或分段TurboID融合构建体应该与适当的标记物共定位,对于一个成功的蛋白质组学实验,TP蛋白的TMT比率应高于FP蛋白,因此ROC图应显示TPR的增长速度快于FPR。绘制TP和FP蛋白质的分布情况也应表明所确定的TMT比率阈值成功地富集了TP蛋白质。例如,在ERM的蛋白质组学图谱绘制中,使用由最大TPR-FPR定义的阈值,ERM蛋白质相对于胞质蛋白质有明显的富集。对于映射已知真阳性较少的区域,如ER-线粒体接触位点,阈值可通过FDR阈值另行确定。蛋白质组学实验产生的候选蛋白质还应该使用独立的方法进行验证,例如免疫印迹,pull-down,功能实验以验证其蛋白相互作用结果是真实可靠的。
总结
这篇文章提供了一个详细的实验协议,使用TurboID和split-TurboID技术在哺乳动物细胞中进行邻近标记,以研究蛋白质-蛋白质相互作用和亚细胞蛋白质组学图谱。通过优化的实验设计和严格的数据分析,该技术能够高分辨率地揭示活细胞内的分子相互作用。文章还讨论了实验中可能遇到的局限性和注意事项,确保了方法的可重复性和结果的可靠性。这项工作不仅推动了生物医学研究的进展,也为疾病机理的探索和新药开发提供了新的工具。
